Analizy genetyczne
Dla bezpieczeństwa zbiorów muzealnych, archiwalnych czy bibliotecznych ogromne znaczenie ma identyfikacja potencjalnego zagrożenia biodeterioracją ze strony mikroorganizmów oraz identyfikacja mikroorganizmów stanowiących potencjalne zagrożenie dla zdrowia ludzi pracujących ze zbiorami.
Obecnie stosowane metody oznaczania liczebności i różnorodności mikroorganizmów zasiedlających badany obiekt zabytkowych opierają się o identyfikację makroskopową, mikroskopową i biochemiczną mikroorganizmów zdolnych do wzrostu na podłożach stałych. Metody te nie dają pełnej informacji dotyczącej wszystkich mikroorganizmów, jakie na badanej powierzchni się znajdują. Takie możliwości dają nam metody identyfikacji mikroorganizmów metodami genetycznymi.
Metody te opierają się o izolację materiału genetycznego i wykorzystanie go, jako matrycy do amplifikacji fragmentu genu 16S rRNA bakterii (region zmienny V3 i V4, około 500 pz) oraz regionu zmiennego ITS1 grzybów i glonów (około 320 pz). Następnie wykonuje się sekwencjonowanie metagenomowe poszczególnych amplikonów z wykorzystaniem technologii Illumina i sekwenatora MiSeq. Uzyskane w wyniku sekwencjonowania dane zostają przeanalizowane z wykorzystaniem specjalistycznego oprogramowania, które pozwala na identyfikację bakterii, grzybów i glonów rzędu nawet kilkudziesięciu tysięcy gatunków w jednej próbce.
Przeprowadzane przez RDLS heritage analizy genetyczne obejmują:
- pobranie i utrwalenie próbek do izolacji DNA
- izolację i oczyszczenie metagenomowego DNA
- przygotowanie amplikonów DNA metod a PCR
- sekwencjonowanie amplikonów
- deponowanie sekwencji w bazach danych
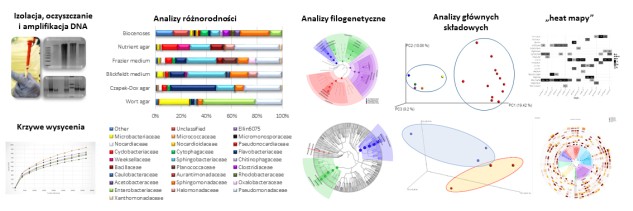
- kompleksową analizę bioinformatyczną uzyskanych sekwencji nukleotydowych:
krzywe wysycenia
analizy różnorodności mikrobiologicznej
analizy głównych składowych
heat maps
W oparciu o sekwencjonowanie metagenomowe możemy przeprowadzić:
1) bezpośrednią identyfikację mikroorganizmów
- z próbek powietrza
- z próbek pobranych z różnych powierzchni
- z próbek o określonej masie, z biofilmów zasiedlających powierzchnie
Efekt końcowy: identyfikacja wszystkich mikroorganizmów obecnych w danej próbce powietrza lub próbce pobranej z powierzchni
2) pośrednią identyfikację mikroorganizmów:
a) wyrosłych na danym typie podłoża mikrobiologicznego.
Sekwencjonowanie przeprowadza się dla materiału genetycznego izolowanego ze wszystkich mikroorganizmów zdolnych do wzrostu na danym podłożu mikrobiologicznym np. na agarze odżywczym czy podłożu Czapek-Doxa. Nie bierzemy pod uwagę typu obiektów zabytkowych, z których pobrano próbki. Dzięki takiej analizie możemy stwierdzić, czy na podłożach dedykowanych dla danej grupy mikroorganizmów obserwujemy wzrost bakterii i/lub grzybów i jakie gatunki dominują na danym podłożu mikrobiologicznym.
Efekt końcowy: identyfikacja mikroorganizmów obecnych w próbce powietrza lub próbce pobranej z powierzchni, zdolnych do tworzenia kolonii na danym typie podłoża mikrobiologicznego – metoda pozwala na identyfikację bakterii i grzybów izolowanych metodami hodowlanymi, charakterystycznych dla danego rodzaju podłoża mikrobiologicznego.
b) wyrosłych na podłożach mikrobiologicznych z próbek pobranych z różnych typów obiektów zabytkowych
Sekwencjonowanie przeprowadza się dla materiału genetycznego izolowanego z mikroorganizmów zdolnych do wzrostu na wszystkich podłożach mikrobiologicznych, na jakie wysiano próbki z danego typu obiektów zabytkowych, np. z rzeźb, mebli czy tkanin. Nie bierzemy pod uwagę rodzaju podłoży mikrobiologicznych, na które wysiano próbki. Dzięki takiej analizie możemy stwierdzić, jakie mikroorganizmy zasiedlają poszczególne rodzaje obiektów zabytkowych i jakie gatunki dominują na danych obiektach.
Efekt końcowy: identyfikacja mikroorganizmów obecnych w próbce pobranej z danego typu obiektów zabytkowych, zdolnych do tworzenia kolonii po wysianiu na podłoża mikrobiologiczne – metoda pozwala na identyfikację bakterii i grzybów charakterystycznych dla danego typu obiektów zabytkowych, izolowanych metodami hodowlanymi.
c) wyrosłych na podłożach mikrobiologicznych z próbek pobranych w różnych pomieszczeniach
Sekwencjonowanie przeprowadza się dla materiału genetycznego izolowanego z mikroorganizmów zdolnych do wzrostu na wszystkich podłożach mikrobiologicznych, na jakie wysiano próbki z różnych typu obiektów zabytkowych pobranych w danym pomieszczeniu lub z próbek powietrza pobranych w danym pomieszczeniu. Nie bierzemy pod uwagę rodzaju podłoży mikrobiologicznych, na które wysiano próbki ani typu obiektów zabytkowych, z których pobrano materiał. Dzięki takiej analizie możemy stwierdzić, jakie mikroorganizmy zasiedlają poszczególne pomieszczenia.
Efekt końcowy: identyfikacja mikroorganizmów obecnych w próbkach pobranych z obiektów zabytkowych lub w próbkach powietrza w danej lokalizacji, zdolnych do tworzenia kolonii po wysianiu na podłoża mikrobiologiczne – metoda pozwala na identyfikację bakterii i grzybów izolowanych metodami hodowlanymi charakterystycznych dla danego pomieszczenia.
d) wyrosłych na podłożach mikrobiologicznych z próbek pobranych z poszczególnych eksponatów
Sekwencjonowanie przeprowadza się dla materiału genetycznego izolowanego z mikroorganizmów zdolnych do wzrostu na wszystkich podłożach mikrobiologicznych, na jakie wysiano próbkę z danego obiektu zabytkowego. Nie bierzemy pod uwagę rodzaju podłoży mikrobiologicznych, na które wysiano próbki. Dzięki takiej analizie możemy stwierdzić, jakie mikroorganizmy zasiedlają poszczególne eksponaty.
Efekt końcowy: identyfikacja mikroorganizmów obecnych w próbkach pobranych z danego eksponatu, zdolnych do tworzenia kolonii po wysianiu na podłoża mikrobiologiczne – metoda pozwala na identyfikację bakterii i grzybów izolowanych metodami hodowlanymi charakterystycznych dla danego eksponatu.
W celu uzyskania bliższych informacji zapraszamy do kontaktu:
heritage@rdls.pl,
tel. 22 554 19 07 lub +48 786 284 496